Capítulo 7 Introdução ao R
Conteudo copiado, com pequenas alterações e atualizações de Capítulo 4 Introdução ao R, do livro Análises Ecológicas no R . O livro está licenciado sob a licença BY-NC-ND 4.0 : https://creativecommons.org/licenses/by-nc-nd/4.0/.
7.1 Contextualização
O objetivo deste capítulo é apresentar os aspectos básicos da linguagem R para a realização dos principais passos para a manipulação, visualização e análise de dados. Abordaremos aqui as questões básicas sobre a linguagem R, como: i) R e RStudio, ii) funcionamento da linguagem, iii) estrutura e manipulação de objetos, iv) exercícios e v) principais livros e material para se aprofundar nos seus estudos.
Todo processo de aprendizagem torna-se mais efetivo quando a teoria é combinada com a prática. Assim, recomendamos fortemente que você, leitor(a) acompanhe os códigos e exercícios deste livro, ao mesmo tempo que os executa em seu computador e não só os leia passivamente. Além disso, se você tiver seus próprios dados é muito importante tentar executar e/ou replicar as análises e/ou gráficos. Por motivos de espaço, não abordaremos todas as questões relacionadas ao uso da linguagem R neste capítulo. Logo, aconselhamos que você consulte o material sugerido no final do capítulo para se aprofundar.
Este capítulo, na maioria das vezes, pode desestimular as pessoas que estão iniciando, uma vez que o mesmo não apresenta os códigos para realizar as análises estatísticas. Contudo, ele é essencial para o entendimento e interpretação do que está sendo informado nas linhas de código, além de facilitar a manipulação dos dados antes de realizar as análises estatísticas. Você perceberá que não usará este capítulo para fazer as análises, mas voltará aqui diversas vezes para relembrar qual é o código ou o que significa determinada expressão ou função usada nos próximos capítulos.
7.2 R e RStudio
Com o R é possível manipular, analisar e visualizar dados, além de escrever desde pequenas linhas de códigos até programas inteiros. O R é a versão em código aberto de uma linguagem de programação chamada de S, criada por John M. Chambers (Stanford University, CA, EUA) nos anos 1980 no Bell Labs. No final dos anos 1990, Robert Gentleman e Ross Ihaka (ambos da Universidade de Auckland, Nova Zelândia), iniciaram o desenvolvimento da versão livre da linguagem S, a linguagem R, com o seguinte histórico: Desenvolvimento (1997-2000), Versão 1 (2000-2004), Versão 2 (2004-2013), Versão 3 (2013-2020) e Versão 4 (2020). Atualmente a linguagem R é mantida por uma rede de colaboradores denominada R Core Team. A origem do nome R é desconhecida, mas reza a lenda que ao lançarem o nome da linguagem os autores se valeram da letra que vinha antes do S, uma vez que a linguagem R foi baseada nela e utilizaram a letra “R”. Outra história conta que pelo fato do nome dos dois autores iniciarem por “R”, batizaram a linguagem com essa letra, vai saber.
Um aspecto digno de nota é que a linguagem R é uma linguagem de programação interpretada, assim como o Python. Isso a faz ser mais fácil de ser utilizada, pois processa linhas de código e as transforma em linguagem de máquina (código binário que o computador efetivamente lê), apesar desse fato diminuir a velocidade de processamento.
Para começarmos a trabalhar com o R é necessário baixá-lo na página do R Project. Os detalhes de instalação são apresentados no Capítulo 6. Reserve um tempo para explorar esta página do R-Project (https://www.r-project.org/). Existem vários livros dedicados a diversos assuntos baseados no R. Além disso, estão disponíveis manuais em diversas línguas para serem baixados gratuitamente.
Como o R é um software livre, não existe a possibilidade de o usuário entrar em contato com um serviço de suporte de usuários. Ao invés disso, existem várias listas de e-mails que fornecem suporte à comunidade de usuários. Nós, particularmente, recomendamos o ingresso nas seguintes listas: R-help, R-sig-ecolog, R-br e discourse.curso-r. Os dois últimos grupos reúnem pessoas usuárias brasileiras do programa R.
Apesar de podermos utilizar o R com o IDE (Ambiente de Desenvolvimento Integrado - Integrated Development Environment) RGui que vem com a instalação da linguagem R para usuários Windows ou no próprio terminal para usuários Linux e MacOS, existem alguns IDEs específicos para facilitar nosso uso dessa linguagem.
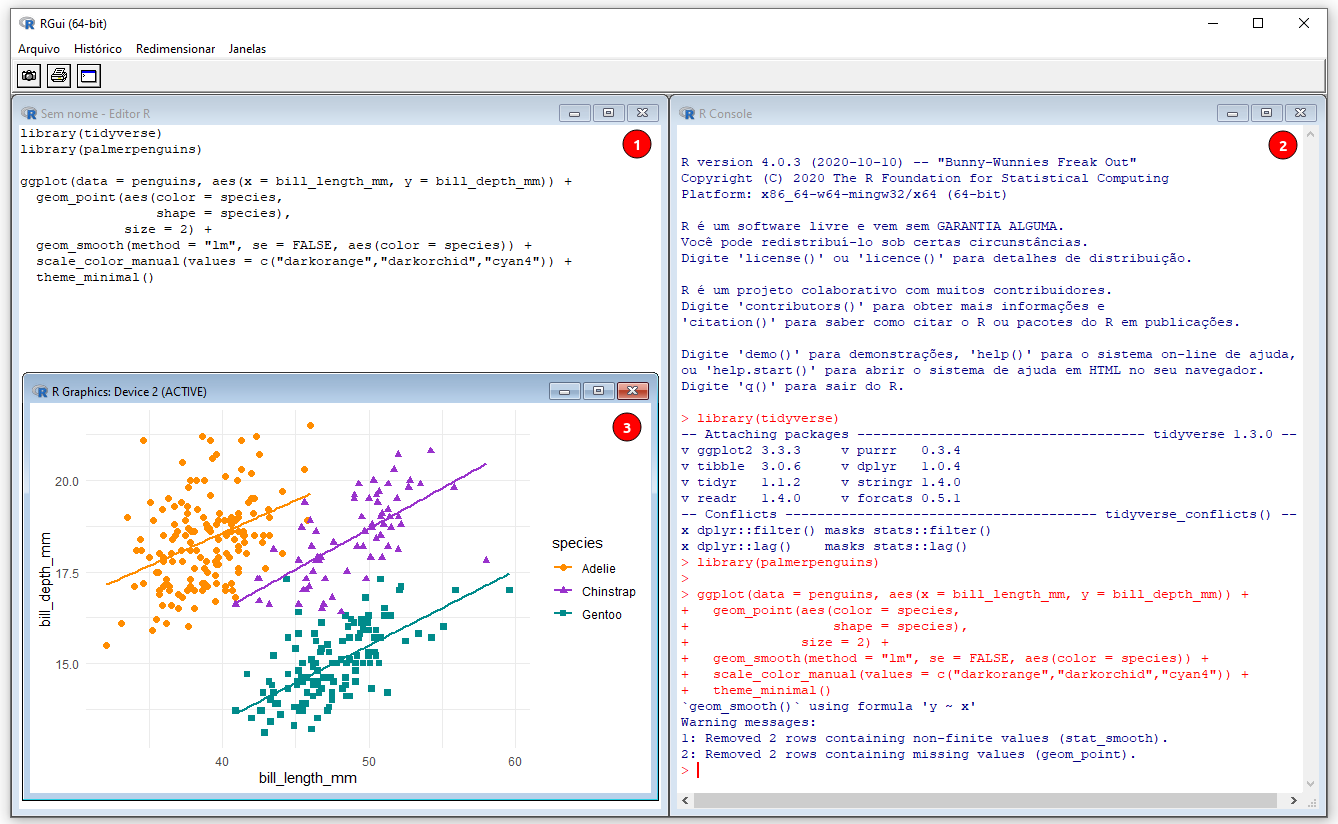
Figura 7.1: Interface do RGui. Os números indicam: (1) R Script, (2) R Console, e (3) R Graphics.
Dessa forma, nós utilizamos o IDE RStudio, e assumimos que você que está lendo fará o mesmo.
O RStudio permite diversas personalizações, grande parte delas contidas em Tools > Global options. Incentivamos as leitoras e leitores a “fuçar” com certa dose de cuidado, nas opções para personalização. Dentre essas mudanças, destacamos três:
Tools > Global options > Appearance > Editor theme: para escolher um tema para seu RStudioTools > Global options > Code > [X] Soft-wrap R source files: com essa opção habilitada, quando escrevemos comentários longos ou mudamos a largura da janela que estamos trabalhando, todo o texto e o código se ajustam a janela automaticamenteTools > Global options > Code > Display > [X Show Margis] e Margin column (80): com essa opção habilitada e para esse valor (80), uma linha vertical irá aparecer no script marcando 80 caracteres, um comprimento máximo recomendado para padronização dos scripts
Importante
Para evitar possíveis erros é importante instalar primeiro o software da linguagem R e depois o IDE RStudio.
O RStudio permite também trabalhar com projetos. Projeto do RStudio é uma forma de organizar os arquivos de scripts e dados dentro de um diretório, facilitando o compartilhamento de fluxo de análises de dados e aumentando assim a reprodutibilidade. Podemos criar um Projeto do RStudio indo em File > New Project ou no ícone de cubo azul escuro que possui um R dentro com um um círculo verde com um sinal de + na parte superior esquerda ou ainda no canto superior direito que possui cubo azul escrito Project que serve para gerenciar os projetos e depois em New Project. Depois de escolher uma dessas opções, uma janela se abrirá onde escolhemos uma das três opções: i) New Directory (para criar um diretório novo com diversas opções), ii) Existing Directory (para escolher um diretório já existente) e iii) Version Control (para criar um projeto que será versionado pelo git ou Subversion).
7.3 Funcionamento da linguagem R
Nesta seção, veremos os principais conceitos para entender como a linguagem R funciona ou como geralmente utilizamos o IDE RStudio no dia a dia, para executar nossas rotinas utilizando a linguagem R. Veremos então: i) console, ii) script, iii) operadores, iv) objetos, v) funções, vi) pacotes, vii) ajuda (help), viii) ambiente (environment/workspace), ix) citações e x) principais erros.
Antes de iniciarmos o uso do R pelo RStudio é fundamental entendermos alguns pontos sobre as janelas e o funcionamento delas no RStudio.
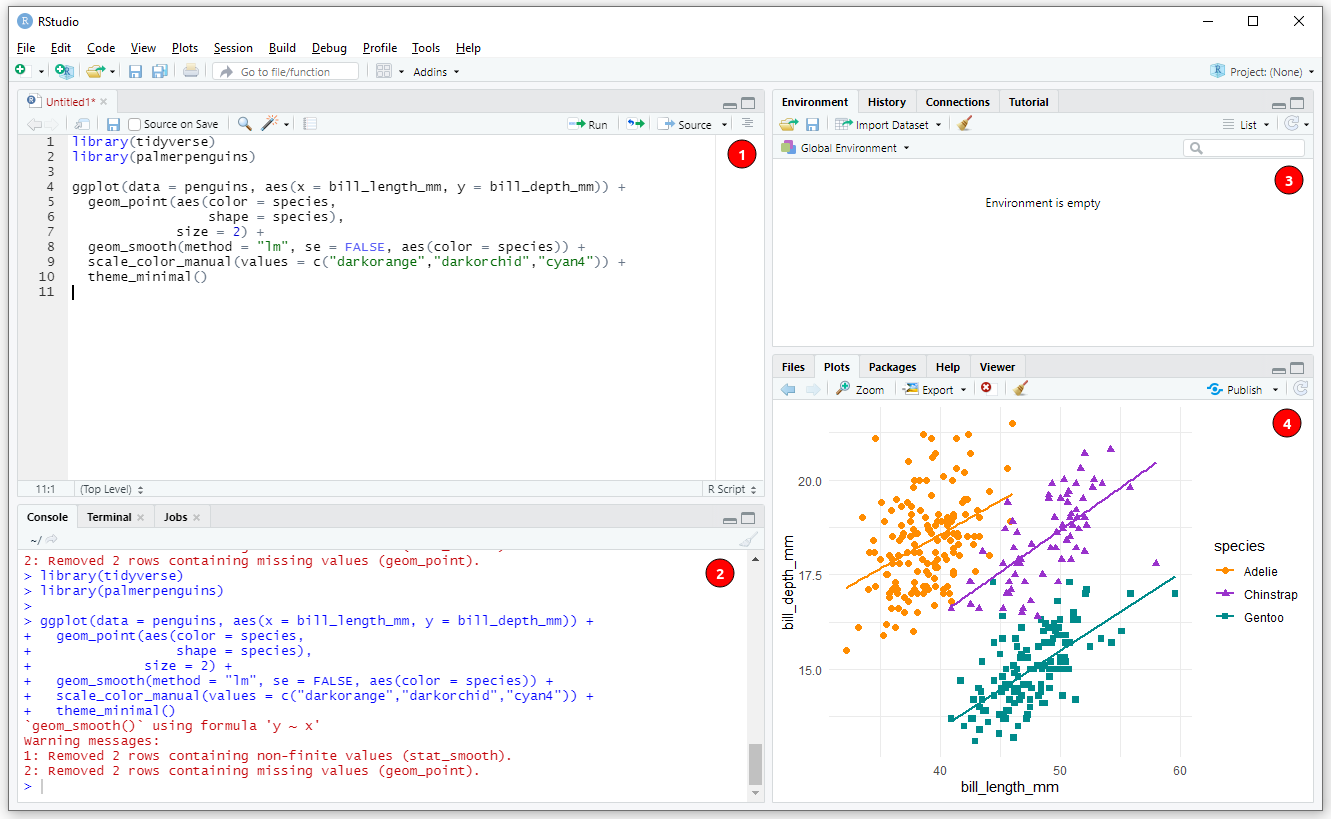
Figura 7.2: Interface do RStudio. Os números indicam: (1) janela com abas de Script, R Markdown, dentre outras; (2) janela com abas de Console, Terminal e Jobs; (3) janela com abas de Environment, History, Conections e Tutorial; e (4) janela com abas de Files, Plots, Packages, Help e Viewer.
Detalhando algumas dessas janelas e abas, temos:
- Console: painel onde os códigos são rodados e vemos as saídas
- Editor/Script: painel onde escrevemos nossos códigos em R, R Markdown ou outro formato
- Environment: painel com todos os objetos criados na sessão
- History: painel com o histórico dos códigos rodados
- Files: painel que mostra os arquivos no diretório de trabalho
- Plots: painel onde os gráficos são apresentados
- Packages: painel que lista os pacotes
- Help: painel onde a documentação das funções é exibida
No RStudio, alguns atalhos são fundamentais para aumentar nossa produtividade:
- F1: abre o painel de Help quando digitado em cima do nome de uma função
- Ctrl + Enter: roda a linha de código selecionada no script
- Ctrl + Shift + N: abre um novo script
- Ctrl + S: salva um script
- Ctrl + Z: desfaz uma operação
- Ctrl + Shift + Z: refaz uma operação
- Alt + -: insere um sinal de atribuição (<-)
- Ctrl + Shift + M: insere um operador pipe (%>%)
- Ctrl + Shift + C: comenta uma linha no script - insere um (#)
- Ctrl + I: indenta (recuo inicial das linhas) as linhas
- Ctrl + Shift + A: reformata o código
- Ctrl + Shift + R: insere uma sessão (# ———————-)
- Ctrl + Shift + H: abre uma janela para selecionar o diretório de trabalho
- Ctrl + Shift + F10: reinicia o console
- Ctrl + L: limpa os códigos do console
- Alt + Shift + K: abre uma janela com todos os atalhos disponíveis
7.3.1 Console
O console é onde a versão da linguagem R instalada é carregada para executar os códigos da linguagem R (Figura 7.2 janela 2). Na janela do console aparecerá o símbolo >, seguido de uma barra vertical | que fica piscando (cursor), onde digitamos ou enviamos nossos códigos do script. Podemos fazer um pequeno exercício: vamos digitar 10 + 2, seguido da tecla Enter para que essa operação seja executada.
## [1] 12O resultado retorna o valor 12, precedido de um valor entre colchetes. Esses colchetes demonstram a posição do elemento numa sequência de valores. Se fizermos essa outra operação 1:42, o R vai criar uma sequência unitária de valores de 1 a 42. A depender da largura da janela do console, vai aparecer um número diferente entre colchetes indicando sua posição na sequência: antes do número 1 vai aparecer o [1], depois quando a sequência for quebrada, vai aparecer o número correspondente da posição do elemento, por exemplo, [37].
## [1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29
## [30] 30 31 32 33 34 35 36 37 38 39 40 41 42Podemos ver o histórico dos códigos executados no console na aba History (Figura 7.2 janela 3).
7.3.2 Scripts
Scripts são arquivos de texto simples, criados com a extensão (terminação) .R (Figura 7.2 janela 1). Para criar um script, basta ir em File > New File > R Script, ou clicar no ícone com uma folha branca e um círculo verde com um sinal de +, logo abaixo de File, ou ainda usando o atalho Ctrl + Shift + N.
Uma vez escrito os códigos no script podemos rodar esses códigos de duas formas: i) todo o script de uma vez, clicando em Source (que fina no canto superior direito da aba script) ou usando o atalho Ctrl + Shift + Enter; ou ii) apenas a linha onde o cursor estiver posicionado, independentemente de sua posição naquela linha, clicando em Run ou usando o atalho Ctrl + Enter.
Devemos sempre salvar nossos scripts, tomando por via de regra: primeiro criar o arquivo e depois ir salvando nesse mesmo arquivo a cada passo de desenvolvimento das análises (não é raro o RStudio fechar sozinho e você perder algum tempo de trabalho). Há diversos motivos para se criar um script: continuar o desenvolvimento desse script em outro momento ou em outro computador, preservar trabalhos passados, ou ainda compartilhar seus códigos com outras pessoas. Para criar ou salvar um script basta ir em File > Save, escolher um diretório e nome para o script e salvá-lo. Podemos ainda utilizar o atalho Ctrl + S.
Em relação aos scripts, há ainda os comentários, representados pelos símbolos # (hash), #' (hash-linha) e #> (hash-maior). A diferença entre eles é que para o segundo e terceiro, quando pressionamos a tecla Enter o comentário #' e #> são inseridos automaticamente na linha seguinte. Linhas de códigos do script contendo comentários em seu início não são lidos pelo console do R. Se o comentário estiver no final da linha, essa linha de código ainda será lida. Os comentários são utilizados geralmente para: i) descrever informações sobre dados ou funções e/ou ii) suprimir linhas de código.
É interessante ter no início de cada script um cabeçalho identificando o objetivo ou análise, autor e data para facilitar o compartilhamento e reprodutibilidade. Os comentários podem ser inseridos ou retirados das linhas com o atalho Ctrl + Shift + C.
#' ---
#' Título: Capítulo 04 - Introdução ao R
#' Autor: Maurício Vancine
#' Data: 11-11-2021
#' ---Além disso, podemos usar comentários para adicionar informações sobre os códigos.
## Comentários
# O R não lê a linha do código depois do # (hash).
42 # Essas palavras não são executadas, apenas o 42, a resposta para questão fundamental da vida, o universo e tudo mais.## [1] 42Por fim, outro ponto fundamental é ter boas práticas de estilo de código. Quanto mais organizado e padronizado estiver seus scripts, mais fácil de entendê-los e de procurar possíveis erros. Existem dois guias de boas práticas para adequar seus scripts: Hadley Wickham e Google. Para simplificar a vida temos o pacote styler (https://styler.r-lib.org/), que serve para adequar o codigo.
Ainda em relação aos scripts, temos os Code Snippets (Fragmentos de código), que são macros de texto usadas para inserir rapidamente fragmentos comuns de código. Por exemplo, o snippet fun insere uma definição de função R. Para mais detalhes, ler o artigo do RStudio Code Snippets.
Uma aplicação bem interessante dos Code Snippets no script é o ts. Basta digitar esse código e em seguida pressionar a tecla Tab para inserir rapidamente a data e horário atuais no script em forma de comentário.
7.3.3 Operadores
No R, podemos agrupar os operadores em cinco tipos: aritméticos, relacionais, lógicos, atribuição e diversos. Grande parte deles são descritos na Tabela “Principais operadores no R.”.
| Operador | Tipo | Descrição |
|---|---|---|
| + | Aritmético | Adição |
| - | Aritmético | Subtração |
| * | Aritmético | Multiplicação |
| / | Aritmético | Divisão |
| %% | Aritmético | Resto da divisão |
| %/% | Aritmético | Divisão inteira |
| ^ ou ** | Aritmético | Expoente |
| > | Relacional | Maior |
| < | Relacional | Menor |
| >= | Relacional | Maior ou igual |
| <= | Relacional | Menor ou igual |
| == | Relacional | Igualdade |
| != | Relacional | Diferença |
| ! | Lógico | Lógico NÃO |
| & | Lógico | Lógico elementar E |
| | | Lógico | Lógico elementar OU |
| && | Lógico | Lógico E |
| || | Lógico | Lógico OU |
| <- ou = | Atribuição | Atribuição à esquerda |
| <<- | Atribuição | Super atribuição à esquerda |
| -> | Atribuição | Atribuição à direita |
| ->> | Atribuição | Super atribuição à direita |
| : | Diversos | Sequência unitária |
| %in% | Diversos | Elementos que pertencem a um vetor |
| %*% | Diversos | Multiplicar matriz com sua transposta |
| %>% | Diversos | Pipe (pacote magrittr) |
| |> | Diversos | Pipe (R base nativo) |
| %–% | Diversos | Intervalo de datas (pacote lubridate) |
Como exemplo, podemos fazer operações simples usando os operadores aritméticos.
## [1] 12## [1] 20Precisamos ficar atentos à prioridade dos operadores aritméticos:
PRIORITÁRIO()>^>* ou />+ ou -NÃO PRIORITÁRIO
Veja no exemplo abaixo como o uso dos parênteses muda o resultado.
## [1] 2.5## [1] 97.3.4 Objetos
Objetos são palavras às quais são atribuídos dados. A atribuição possibilita a manipulação de dados ou armazenamento dos resultados de análises. Utilizaremos os símbolos < (menor), seguido de - (menos), sem espaço, dessa forma <-. Também podemos utilizar o símbolo de igual (=), mas não recomendamos, por não fazer parte das boas práticas de escrita de códigos em R. Podemos inserir essa combinação de símbolos com o atalho Alt + -. Para demonstrar, vamos atribuir o valor 10 à palavra obj_10, e chamar esse objeto novamente para verificar seu conteúdo.
## [1] 10 Importante
Recomendamos sempre verificar o conteúdo dos objetos chamando-os novamente para confirmar se a atribuição foi realizada corretamente e se o conteúdo corresponde à operação realizada.
Todos os objetos criados numa sessão do R ficam listados na aba Environment . Além disso, o RStudio possui a função autocomplete, ou seja, podemos digitar as primeiras letras de um objeto (ou função) e em seguida apertar Tab para que o RStudio liste tudo que começar com essas letras.
Dois pontos importantes sobre atribuições: primeiro, o R sobrescreve os valores dos objetos com o mesmo nome, deixando o objeto com o valor da última atribuição.
## [1] 100## [1] 2Segundo, o R tem limitações ao nomear objetos:
- nome de objetos só podem começar por letras (
a-zouA-Z) ou pontos (.) - nome de objetos só podem conter letras (
a-zouA-Z), números (0-9), underscores (_) ou pontos (.) - R é case-sensitive, i.e., ele reconhece letras maiúsculas como diferentes de letras minúsculas. Assim, um objeto chamado “resposta” é diferente do objeto “RESPOSTA”
- devemos evitar acentos ou cedilha (
ç) para facilitar a memorização dos objetos e também para evitar erros de codificação (encoding) de caracteres - nomes de objetos não podem ser iguais a nomes especiais, reservados para programação (
break,else,FALSE,for,function,if,Inf,NA,NaN,next,repeat,return,TRUE,while)
Podemos ainda utilizar objetos para fazer operações e criar objetos. Isso pode parecer um pouco confuso para os iniciantes, mas é fundamental aprender essa lógica para passar para os próximos passos.
## Definir dois objetos
va1 <- 10
va2 <- 2
## Operações com objetos e atribuicão
adi <- va1 + va2
adi## [1] 127.3.5 Funções
Funções são códigos preparados para realizar uma tarefa específica de modo simples. Outra forma de entender uma função é: códigos que realizam operações em argumentos. Devemos retomar ao conceito do ensino médio de funções: os dados de entrada são argumentos e a função realizará alguma operação para modificar esses dados de entrada. A estrutura de uma função é muito similar à sintaxe usada em planilhas eletrônicas, sendo composta por:
nome_da_função(argumento1, argumento2, …)
- Nome da função: remete ao que ela faz
- Parênteses: limitam a função
- Argumentos: valores, parâmetros ou expressões onde a função atuará
- Vírgulas: separam os argumentos
Os argumentos de uma função podem ser de dois tipos:
- Valores ou objetos: a função alterará os valores em si ou os valores atribuídos aos objetos
- Parâmetros: valores fixos que informam um método ou a realização de uma operação. Informa-se o nome desse argumento, seguido de “=” e um número, texto ou TRUE ou FALSE
Alguns exemplos de argumentos como valores ou objetos.
## [1] 12## [1] 12Vamos ver agora alguns exemplos de argumentos usados como parâmetros. Note que apesar do valor do argumento ser o mesmo (10), seu efeito no resultado da função rep() muda drasticamente. Aqui também é importante destacar um ponto: i) podemos informar os argumentos sequencialmente, sem explicitar seus nomes, ou ii) independente da ordem, mas explicitando seus nomes. Entretanto, como no exemplo abaixo, devemos informar o nome do argumento (i.e., parâmetro), para que seu efeito seja o que desejamos.
## Funções - argumentos como parâmetros
## Repetição - repete todos os elementos
rep(x = 1:5, times = 10)## [1] 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5 1 2 3 4
## [45] 5 1 2 3 4 5## [1] 1 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2 2 3 3 3 3 3 3 3 3 3 3 4 4 4 4 4 4 4 4 4 4 5 5 5 5
## [45] 5 5 5 5 5 5Um ponto fundamental e que deve ser entendido é o fluxo de atribuições do resultado da operação de funções a novos objetos. No desenvolvimento de qualquer script na linguagem R, grande parte da estrutura do mesmo será dessa forma: atribuição de dados a objetos > operações com funções > atribuição dos resultados a novos objetos > operações com funções desses novos objetos > atribuição dos resultados a novos objetos. Ao entender esse funcionamento, começamos a entender como devemos pensar na organização do nosso script para montar as análises que precisamos.
## [1] 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5 1 2 3 4 5 1 2 3 4
## [45] 5 1 2 3 4 5## [1] 150## [1] 12.24745Por fim, é fundamental também entender a origem das funções que usamos no R. Todas as funções são advindas de pacotes. Esses pacotes possuem duas origens.
- pacotes já instalados por padrão e que são carregados quando abrimos o R (R Base)
- pacotes que instalamos e carregamos com funções
7.3.6 Pacotes
Pacotes são conjuntos extras de funções para executar tarefas específicas, além dos pacotes instalados no R Base. Existe literalmente milhares de pacotes (~19,000 enquanto estamos escrevendo esse livro) para as mais diversas tarefas: estatística, ecologia, geografia, sensoriamento remoto, econometria, ciências sociais, gráficos, machine learning, etc. Podemos verificar este vasto conjunto de pacotes pelo link que lista por nome os pacotes oficiais, ou seja, que passaram pelo crivo do CRAN. Existem ainda muito mais pacotes em desenvolvimento, geralmente disponibilizados em repositórios do GitHub ou GitLab.
Podemos listar esses pacotes disponíveis no CRAN com esse código.
Primeiramente, com uma sessão do R sem carregar nenhum pacote extra, podemos verificar pacotes carregados pelo R Base utilizando a função search().
Podemos ainda verificar todos pacotes instalados em nosso computador com a função library().
No R, quando tratamos de pacotes, devemos destacar a diferença de dois conceitos: instalar um pacote e carregar um pacote. A instalação de pacotes possui algumas características:
- Instala-se um pacote apenas uma vez
- Precisamos estar conectados à internet
- O nome do pacote precisa estar entre aspas na função de instalação
- Função (CRAN):
install.packages()
Vamos instalar o pacote vegan diretamente do CRAN, que possui funções para realizar uma série de análise em ecologia. Para isso, podemos ir em Tools > Install Packages..., ou ir na aba Packages , procurar o pacote e simplesmente clicar em “Install”. Podemos ainda utilizar a função install.packages().
Podemos conferir em que diretórios um pacote será instalado com a função .libPaths().
## [1] "C:/Users/user/AppData/Local/R/win-library/4.3"
## [2] "C:/Program Files/R/R-4.3.2/library" Importante
Uma vez instalado um pacote, não há necessidade de instalá-lo novamente. Entretanto, todas às vezes que iniciarmos uma sessão no R, precisamos carregar os pacotes com as funções que precisamos utilizar.
O carregamento de pacotes possui algumas características:
- Carrega-se o pacote toda vez que se abre uma nova sessão do R
- Não precisamos estar conectados à internet
- O nome do pacote não precisa estar entre aspas na função de carregamento
- Funções:
library()ourequire()
Vamos carregar o pacote vegan que instalamos anteriormente. Podemos ir na aba Packages e assinalar o pacote que queremos carregar ou utilizar a função library().
Como dissemos, alguns pacotes em desenvolvimento encontram-se disponíveis em repositórios como por exemplo: GitHub, GitLab e Bioconductor. Para instalar pacotes do GitHub, por exemplo, precisamos instalar e carregar o pacote remotes. Para funcionar, deve instalar “RTools” antes (https://cran.r-project.org/bin/windows/Rtools/) .
Uma vez instalado e carregado esse pacote, podemos instalar o pacote do GitHub, utilizando a função remotes::install_github(). Precisamos atentar para usar essa forma “nome_usuario/nome_repositorio”, retirados do link do repositório de interesse. Como exemplo, podemos instalar o pacote eprdados do repositório do GitHub darrennorris/eprdados e depois utilizar a função library() para carregá-lo. Para funcionar, deve instalar “RTools” antes (https://cran.r-project.org/bin/windows/Rtools/) .
## Instalar pacote do github
remotes::install_github("darrennorris/eprdados")
## Carregar pacote do github
library("eprdados")Podemos ver a descrição de um pacote com a função packageDescription().
## Package: vegan
## Title: Community Ecology Package
## Version: 2.6-4
## Authors@R: c(person("Jari", "Oksanen", role=c("aut","cre"),
## email="jhoksane@gmail.com"), person("Gavin L.", "Simpson", role="aut",
## email="ucfagls@gmail.com"), person("F. Guillaume", "Blanchet",
## role="aut"), person("Roeland", "Kindt", role="aut"), person("Pierre",
## "Legendre", role="aut"), person("Peter R.", "Minchin", role="aut"),
## person("R.B.", "O'Hara", role="aut"), person("Peter", "Solymos",
## role="aut"), person("M. Henry H.", "Stevens", role="aut"),
## person("Eduard", "Szoecs", role="aut"), person("Helene", "Wagner",
## role="aut"), person("Matt", "Barbour", role="aut"), person("Michael",
## "Bedward", role="aut"), person("Ben", "Bolker", role="aut"),
## person("Daniel", "Borcard", role="aut"), person("Gustavo", "Carvalho",
## role="aut"), person("Michael", "Chirico", role="aut"), person("Miquel",
## "De Caceres", role="aut"), person("Sebastien", "Durand", role="aut"),
## person("Heloisa Beatriz Antoniazi", "Evangelista", role="aut"),
## person("Rich", "FitzJohn", role="aut"), person("Michael", "Friendly",
## role="aut"), person("Brendan","Furneaux", role="aut"),
## person("Geoffrey", "Hannigan", role="aut"), person("Mark O.", "Hill",
## role="aut"), person("Leo", "Lahti", role="aut"), person("Dan",
## "McGlinn", role="aut"), person("Marie-Helene", "Ouellette", role="aut"),
## person("Eduardo", "Ribeiro Cunha", role="aut"), person("Tyler", "Smith",
## role="aut"), person("Adrian", "Stier", role="aut"), person("Cajo J.F.",
## "Ter Braak", role="aut"), person("James", "Weedon", role="aut"))
## Depends: permute (>= 0.9-0), lattice, R (>= 3.4.0)
## Suggests: parallel, tcltk, knitr, markdown
## Imports: MASS, cluster, mgcv
## VignetteBuilder: utils, knitr
## Description: Ordination methods, diversity analysis and other functions for
## community and vegetation ecologists.
## License: GPL-2
## BugReports: https://github.com/vegandevs/vegan/issues
## URL: https://github.com/vegandevs/vegan
## NeedsCompilation: yes
## Packaged: 2022-10-11 08:36:07 UTC; jarioksa
## Author: Jari Oksanen [aut, cre], Gavin L. Simpson [aut], F. Guillaume Blanchet
## [aut], Roeland Kindt [aut], Pierre Legendre [aut], Peter R. Minchin
## [aut], R.B. O'Hara [aut], Peter Solymos [aut], M. Henry H. Stevens
## [aut], Eduard Szoecs [aut], Helene Wagner [aut], Matt Barbour [aut],
## Michael Bedward [aut], Ben Bolker [aut], Daniel Borcard [aut], Gustavo
## Carvalho [aut], Michael Chirico [aut], Miquel De Caceres [aut],
## Sebastien Durand [aut], Heloisa Beatriz Antoniazi Evangelista [aut],
## Rich FitzJohn [aut], Michael Friendly [aut], Brendan Furneaux [aut],
## Geoffrey Hannigan [aut], Mark O. Hill [aut], Leo Lahti [aut], Dan
## McGlinn [aut], Marie-Helene Ouellette [aut], Eduardo Ribeiro Cunha
## [aut], Tyler Smith [aut], Adrian Stier [aut], Cajo J.F. Ter Braak [aut],
## James Weedon [aut]
## Maintainer: Jari Oksanen <jhoksane@gmail.com>
## Repository: CRAN
## Date/Publication: 2022-10-11 12:40:02 UTC
## Built: R 4.3.2; x86_64-w64-mingw32; 2023-11-02 03:07:22 UTC; windows
## Archs: x64
##
## -- File: C:/Users/user/AppData/Local/R/win-library/4.3/vegan/Meta/package.rdsA maioria dos pacotes possui conjuntos de dados que podem ser acessados pela função data(). Esses conjuntos de dados podem ser usados para testar as funções do pacote. Se estiver com dúvida na maneira como você deve preparar a planilha para realizar uma análise específica, entre na Ajuda (Help) da função e veja os conjuntos de dados que estão no exemplo desta função. Como exemplo, vamos carregar os dados dune do pacote vegan, que são dados de observações de 30 espécies vegetais em 20 locais.
## Achimill Agrostol Airaprae Alopgeni Anthodor Bellpere
## 1 1 0 0 0 0 0
## 2 3 0 0 2 0 3
## 3 0 4 0 7 0 2
## 4 0 8 0 2 0 2
## 5 2 0 0 0 4 2
## 6 2 0 0 0 3 0E um último ponto fundamental sobre pacotes, diz respeito à atualização dos mesmos. Os pacotes são atualizados com frequência, e infelizmente (ou felizmente, pois as atualizações podem oferecer algumas quebras entre pacotes), não se atualizam sozinhos. Muitas vezes, a instalação de um pacote pode depender da versão dos pacotes dependentes, e geralmente uma janela com diversas opções numéricas se abre perguntando se você quer que todos os pacotes dependentes sejam atualizados. Podemos ir na aba Packages e clicar em “Update” ou usar a função update.packages(checkBuilt = TRUE, ask = FALSE) para atualizá-los, entretanto, essa é uma função que costuma demorar muito para terminar de ser executada.
Destacamos e incentivamos ainda uma prática que achamos interessante para aumentar a reprodutibilidade de nossos códigos e scripts: a de chamar as funções de pacotes carregados dessa forma pacote::função(). Com o uso dessa prática, deixamos claro o pacote em que a função está implementada. Esta prática é importante por que com frequência pacotes diferentes criam funções com mesmo nome, mas com características internas (argumentos) diferentes. Assim, não expressar o pacote de interesse pode gerar erros na execução de suas análises. Destacamos aqui o exemplo de como instalar pacotes do GitHub do pacote remotes.
7.3.7 Ajuda (Help)
Um importante passo para melhorar a usabilidade e ter mais familiaridade com a linguagem R é aprender a usar a ajuda (help) de cada função. Para tanto, podemos utilizar a função help() ou o operador ?, depois de ter carregado o pacote. O arquivo de ajuda do R possui tópicos, que nos auxiliam muito no entendimento dos dados de entrada, argumentos e que operações estão sendo realizadas.Abaixo descrevemos esses tópicos:
- Description: resumo da função
- Usage: como utilizar a função e quais os seus argumentos
- Arguments: detalha os argumentos e como os mesmos devem ser especificados
- Details: detalhes importantes para se usar a função
- Value: mostra como interpretar a saída (output) da função (os resultados)
- Note: notas gerais sobre a função
- Authors: autores da função
- References: referências bibliográficas para os métodos usados para construção da função
- See also: funções relacionadas
- Examples: exemplos do uso da função. Às vezes pode ser útil copiar esse trecho e colar no R para ver como funciona e como usar a função.
Vamos realizar um exemplo, buscando o help da função aov(), que realiza uma análise de variância.
Além das funções, podemos buscar detalhes de um pacote específico, para uma página simples do help utilizando a função help() ou o operador ?. Entretanto, para uma opção que ofereça uma descrição detalhada e um índice de todas as funções do pacote, podemos utilizar a função library(), mas agora utilizando o argumento help, indicando o pacote de interesse entre aspas.
Podemos ainda procurar o nome de uma função para realizar uma análise específica utilizando a função help.search() com o termo que queremos em inglês e entre aspas.
Outra ferramenta de busca é a página rseek, na qual é possível buscar por um termo não só nos pacotes do R, mas também em listas de emails, manuais, páginas na internet e livros sobre o programa.
7.3.8 Ambiente (Environment)
O ambiente (environment), como vimos, é onde os objetos criados são armazenados. É fundamental entender que um objeto é uma alocação de um pequeno espaço na memória RAM do nosso computador, onde o R armazenará um valor ou o resultado de uma função, utilizando o nome dos objetos que definimos na atribuição. Sendo assim, se fizermos a atribuição de um objeto maior que o tamanho da memória RAM do nosso computador, esse objeto não será alocado, e a atribuição não funcionará, retornando um erro. Existem opções para contornar esse tipo de limitação, mas não a abordaremos aqui. Entretanto, podemos utilizar a função object.size() para saber quanto espaço nosso objeto criado está alocando de memória RAM.
## 56 bytesPodemos listar todos os objetos criados com a função ls() ou objects().
Podemos ainda remover todos os objetos criados com a função rm() ou remove(). Ou ainda fazer uma função composta para remover todos os objetos do Environment.
Quando usamos a função ls() agora, nenhum objeto é listado.
## character(0)Toda a vez que fechamos o R os objetos criados são apagados do Environment. Dessa forma, em algumas ocasiões, por exemplo, análises estatísticas que demoram um grande tempo para serem realizadas, pode ser interessante exportar alguns ou todos os objetos criados.
Para salvar todos os objetos, ou seja, todo o Workspace, podemos ir em Session -> Save Workspace As... e escolher o nome do arquivo do Workspace, por exemplo, “meu_workspace.RData”. Podemos ainda utilizar funções para essas tarefas. A função save.image() salva todo Workspace com a extensão .RData.
Depois disso, podemos fechar o RStudio tranquilamente e quando formos trabalhar novamente, podemos carregar os objetos criados indo em Session -> Load Workspace... ou utilizando a função load().
Entretanto, em algumas ocasiões, não precisamos salvar todos os objetos. Dessa forma, podemos salvar apenas alguns objetos específicos usando a função save(), também com a extensão .RData.
## Salvar apenas um objeto
save(obj1, file = "meu_obj.RData")
## Salvar apenas um objeto
save(obj1, obj2, file = "meus_objs.RData")
## Carregar os objetos
load("meus_objs.RData")Ou ainda, podemos salvar apenas um objeto com a extensão .rds. Para isso, usamos as funções saveRDS() e readRDS(), para exportar e importar esses dados, respectivamente. É importante ressaltar que nesse formato .rds, apenas um objeto é salvo por arquivo criado e que para que o objeto seja criado no Workspace do R, ele precisa ser lido e atribuído à um objeto.
7.3.9 Citações
Ao utilizar o R para realizar alguma análise em nossos estudos, é fundamental a citação do mesmo. Para saber como citar o R em artigos, existe uma função denominada citation(), que provê um formato genérico de citação e um BibTeX para arquivos LaTeX e R Markdown.
## To cite R in publications use:
##
## R Core Team (2023). _R: A Language and Environment for Statistical Computing_.
## R Foundation for Statistical Computing, Vienna, Austria.
## <https://www.R-project.org/>.
##
## A BibTeX entry for LaTeX users is
##
## @Manual{,
## title = {R: A Language and Environment for Statistical Computing},
## author = {{R Core Team}},
## organization = {R Foundation for Statistical Computing},
## address = {Vienna, Austria},
## year = {2023},
## url = {https://www.R-project.org/},
## }
##
## We have invested a lot of time and effort in creating R, please cite it when
## using it for data analysis. See also 'citation("pkgname")' for citing R packages.No resultado dessa função, há uma mensagem muito interessante: “See also ‘citation(“pkgname”)’ for citing R packages.”. Dessa forma, aconselhamos, sempre que possível, claro, citar também os pacotes utilizados nas análises para dar os devidos créditos aos desenvolvedores e desenvolvedoras das funções implementadas nos pacotes. Como exemplo, vamos ver como fica a citação do pacote vegan.
## To cite package 'vegan' in publications use:
##
## Oksanen J, Simpson G, Blanchet F, Kindt R, Legendre P, Minchin P, O'Hara R,
## Solymos P, Stevens M, Szoecs E, Wagner H, Barbour M, Bedward M, Bolker B,
## Borcard D, Carvalho G, Chirico M, De Caceres M, Durand S, Evangelista H,
## FitzJohn R, Friendly M, Furneaux B, Hannigan G, Hill M, Lahti L, McGlinn D,
## Ouellette M, Ribeiro Cunha E, Smith T, Stier A, Ter Braak C, Weedon J (2022).
## _vegan: Community Ecology Package_. R package version 2.6-4,
## <https://CRAN.R-project.org/package=vegan>.
##
## A BibTeX entry for LaTeX users is
##
## @Manual{,
## title = {vegan: Community Ecology Package},
## author = {Jari Oksanen and Gavin L. Simpson and F. Guillaume Blanchet and Roeland Kindt and Pierre Legendre and Peter R. Minchin and R.B. O'Hara and Peter Solymos and M. Henry H. Stevens and Eduard Szoecs and Helene Wagner and Matt Barbour and Michael Bedward and Ben Bolker and Daniel Borcard and Gustavo Carvalho and Michael Chirico and Miquel {De Caceres} and Sebastien Durand and Heloisa Beatriz Antoniazi Evangelista and Rich FitzJohn and Michael Friendly and Brendan Furneaux and Geoffrey Hannigan and Mark O. Hill and Leo Lahti and Dan McGlinn and Marie-Helene Ouellette and Eduardo {Ribeiro Cunha} and Tyler Smith and Adrian Stier and Cajo J.F. {Ter Braak} and James Weedon},
## year = {2022},
## note = {R package version 2.6-4},
## url = {https://CRAN.R-project.org/package=vegan},
## }Podemos ainda utilizar a função write_bib() do pacote knitr para exportar a citação do pacote no formato .bib.
7.3.10 Principais erros de iniciantes

Errar quando se está começando a usar o R é muito comum e faz parte do aprendizado. Entretanto, os erros nunca devem ser encarados como uma forma de desestímulo, mas sim como um desafio para continuar tentando. Todos nós, autores deste livro inclusive, e provavelmente usuários mais ou menos experientes, já passaram por um momento em que se quer desistir de tudo. Jovem aprendiz de R, a única diferença entre você que está iniciando agora e nós que usamos o R há mais tempo são as horas a mais de uso (e ódio). O que temos a mais é experiência para olhar o erro, lê-lo e conseguir interpretar o que está errado e saber buscar ajuda.
Dessa forma, o ponto mais importante de quem está iniciando é ter paciência, calma, bom humor, ler e entender as mensagens de erros. Recomendamos uma prática que pode ajudar: caso não esteja conseguindo resolver alguma parte do seu código, deixe ele de lado um tempo, descanse, faça uma caminhada, tome um banho, converse com seus animais de estimação ou plantas, tenha um pato de borracha ou outro objeto inanimado (um dos autores tem um sapinho de madeira), explique esse código para esse pato (processo conhecido como Debug com Pato de Borracha), logo a solução deve aparecer.
Listaremos aqui o que consideramos os principais erros dos iniciantes no R.
1. Esquecer de completar uma função ou bloco de códigos
Esquecer de completar uma função ou bloco de códigos é algo bem comum. Geralmente esquecemos de fechar aspas "" ou parênteses (), mas geralmente o R nos informa isso, indicando um símbolo de + no console. Se você cometeu esse erro, lembre-se de apertar a tecla esc do seu computador clicando antes com o cursor do mouse no console do R.
## Error: <text>:3:0: unexpected end of input
## 1: sum(1, 2
## 2: +
## ^2. Esquecer de vírgulas dentro de funções
Outro erro bastante comum é esquecer de acrescentar a vírgula , para separar argumentos dentro de uma função, principalmente se estamos compondo várias funções acopladas, i.e., uma função dentro da outra.
## Error: <text>:1:7: unexpected numeric constant
## 1: sum(1 2
## ^3. Chamar um objeto pelo nome errado
Pode parecer simples, mas esse é de longe o erro mais comum que pessoas iniciantes comentem. Quando temos um script longo, é de se esperar que tenhamos atribuído diversos objetos e em algum momento atribuímos um nome do qual não lembramos. Dessa forma, quando chamamos o objeto ele não existe e o console informa um erro. Entretanto, esse tipo de erro pode ser facilmente identificado, como o exemplo abaixo.
## Error in eval(expr, envir, enclos): object 'OBJ' not found4. Esquecer de carregar um pacote
Esse também é um erro recorrente, mesmo para usuários mais experientes. Em scripts de análises complexas, que requerem vários pacotes, geralmente esquecemos de um ou outro pacote. A melhor forma de evitar esse tipo de erro é listar os pacotes que vamos precisar usar logo no início do script.
## Warning in data(dune): data set 'dune' not found## Error in decostand(dune[1:6, 1:6], "hell"): could not find function "decostand"Geralmente a mensagem de erro será de que a função não foi encontrada ou algo nesse sentido. Carregando o pacote, esse erro é contornado.
## This is vegan 2.6-4##
## Attaching package: 'vegan'## The following object is masked from 'package:parsnip':
##
## nullmodel## Achimill Agrostol Airaprae Alopgeni Anthodor Bellpere
## 1 1.0000000 0.0000000 0 0.0000000 0.0000000 0.0000000
## 2 0.6123724 0.0000000 0 0.5000000 0.0000000 0.6123724
## 3 0.0000000 0.5547002 0 0.7337994 0.0000000 0.3922323
## 4 0.0000000 0.8164966 0 0.4082483 0.0000000 0.4082483
## 5 0.5000000 0.0000000 0 0.0000000 0.7071068 0.5000000
## 6 0.6324555 0.0000000 0 0.0000000 0.7745967 0.00000005. Usar o nome da função de forma errônea
Esse erro não é tão comum, mas pode ser incômodo às vezes. Algumas funções possuem nomes no padrão “Camel Case”, i.e., com letras maiúsculas no meio do nome da função. Isso às vezes pode confundir, ou ainda, as funções podem ou não ser separadas com ., como row.names() e rownames().
## Error in colsums(dune): could not find function "colsums"## Achimill Agrostol Airaprae Alopgeni Anthodor Bellpere Bromhord Chenalbu Cirsarve Comapalu
## 16 48 5 36 21 13 15 1 2 4
## Eleopalu Elymrepe Empenigr Hyporadi Juncarti Juncbufo Lolipere Planlanc Poaprat Poatriv
## 25 26 2 9 18 13 58 26 48 63
## Ranuflam Rumeacet Sagiproc Salirepe Scorautu Trifprat Trifrepe Vicilath Bracruta Callcusp
## 14 18 20 11 54 9 47 4 49 106. Atentar para o diretório correto
Muitas vezes o erro é simplesmente porque o usuário(a) não definiu o diretório correto onde está o arquivo a ser importado ou exportado. Por isso é fundamental sempre verificar se o diretório foi definido corretamente, geralmente usando as funções dir() ou list.files() para listar no console a lista de arquivos no diretório. Podemos ainda usar o argumento pattern para listar arquivos por um padrão textual.
## Listar os arquivos do diretório definido
dir()
list.files()
## Listar os arquivos do diretório definido por um padrão
dir(pattern = ".csv")Além disso, é fundamental ressaltar a importância de verificar se o nome do arquivo que importaremos foi digitado corretamente, atentando-se também para a extensão: .csv, .txt, .xlsx, etc.
7.4 Estrutura e manipulação de objetos
O conhecimento sobre a estrutura e manipulação de objetos é fundamental para ter domínio e entendimento do funcionamento da linguagem R. Nesta seção, trataremos da estrutura e manipulação de dados no R, no que ficou conhecido como modo R Base, em contrapartida ao tidyverse, tópico tratado no Capítulo 8. Abordaremos aqui temas chaves, como: i) atributos de objetos, ii) manipulação de objetos unidimensionais e multidimensionais, iii) valores faltantes e especiais, iv) diretório de trabalho e v) importar, conferir e exportar dados tabulares.
7.4.1 Atributo dos objetos
Quando fazemos atribuições de dados no R (<-), os objetos gerados possuem três características.
- Nome: palavra que o R reconhece os dados atribuídos
- Conteúdo: dados em si
- Atributos: modos (natureza) e estruturas (organização) dos elementos
Vamos explorar mais a fundo os modos e estruturas dos objetos. Vale ressaltar que isso é uma simplificação, pois há muitas classes de objetos, como funções e saídas de funções que possuem outros atributos.
Podemos verificar os atributos dos objetos com a função attributes().
## $names
## [1] "Achimill" "Agrostol" "Airaprae" "Alopgeni" "Anthodor" "Bellpere" "Bromhord" "Chenalbu"
## [9] "Cirsarve" "Comapalu" "Eleopalu" "Elymrepe" "Empenigr" "Hyporadi" "Juncarti" "Juncbufo"
## [17] "Lolipere" "Planlanc" "Poaprat" "Poatriv" "Ranuflam" "Rumeacet" "Sagiproc" "Salirepe"
## [25] "Scorautu" "Trifprat" "Trifrepe" "Vicilath" "Bracruta" "Callcusp"
##
## $row.names
## [1] "1" "2" "3" "4" "5" "6" "7" "8" "9" "10" "11" "12" "13" "14" "15" "16" "17"
## [18] "18" "19" "20"
##
## $class
## [1] "data.frame"Modo dos objetos
A depender da natureza dos elementos que compõem os dados e que foram atribuídos aos objetos, esses objetos podem ser, de forma simples um dos cinco modos: numérico do tipo inteiro (integer), numérico do tipo flutuante (double), texto (character), lógico (logical) ou complexo (complex).
A atribuição de números no R pode gerar dois tipos de modos: integer para números inteiros e double para números flutuantes ou com decimais.
## [1] "numeric"## [1] "double"A título de praticidade, ambos são incorporados como o modo numeric, com o tipo double, a menos que especifiquemos que seja inteiro com a letra L depois do número, representando a palavra Larger, geralmente usando para armazenar números muito grandes.
## [1] "numeric"## [1] "integer"Além de números, podemos atribuir textos, utilizando para isso aspas "".
## [1] "character"Em algumas situações, precisamos indicar a ocorrência ou não de um evento ou uma operação. Para isso, utilizamos as palavras reservadas (TRUE e FALSE), chamadas de variáveis booleanas, pois assumem apenas duas possibilidades: falso (0) ou verdadeiro (1). Devemos nos ater para o fato dessas palavras serem escritas com letras maiúsculas e sem aspas.
## [1] "logical"Por fim, existe um modo pouco utilizado que cria números complexos (raiz de números negativos).
## [1] "complex"Podemos verificar o modo dos objetos ou fazer a conversão entre esses modos com diversas funções.
## Verificar o modo dos objetos
is.numeric()
is.integer()
is.character()
is.logical()
is.complex()
## Conversões entre modos
as.numeric()
as.integer()
as.character()
as.logical()
as.complex()
## Exemplo
num <- 1:5
num
mode(num)
cha <- as.character(num)
cha
mode(cha)Estrutura dos objetos
Uma vez entendido a natureza dos modos dos elementos dos objetos no R, podemos passar para o passo seguinte e entender como esses elementos são estruturados dentro dos objetos.
Essa estruturação irá nos contar sobre a organização dos elementos, com relação aos modos e dimensionalidade da disposição desses elementos. De modo bem simples, os elementos podem ser estruturados em cinco tipos:
- Vetores e fatores: homogêneo (um modo) e unidimensional (uma dimensão). Um tipo especial de vetor são os fatores, usados para designar variáveis categóricas
- Matrizes: homogêneo (um modo) e bidimensional (duas dimensões)
- Arrays: homogêneo (um modo) e multidimensional (mais de duas dimensões)
- Data frames: heterogêneo (mais de um modo) e bidimensional (duas dimensões)
- Listas: heterogêneo (mais de um modo) e unidimensional (uma dimensão)
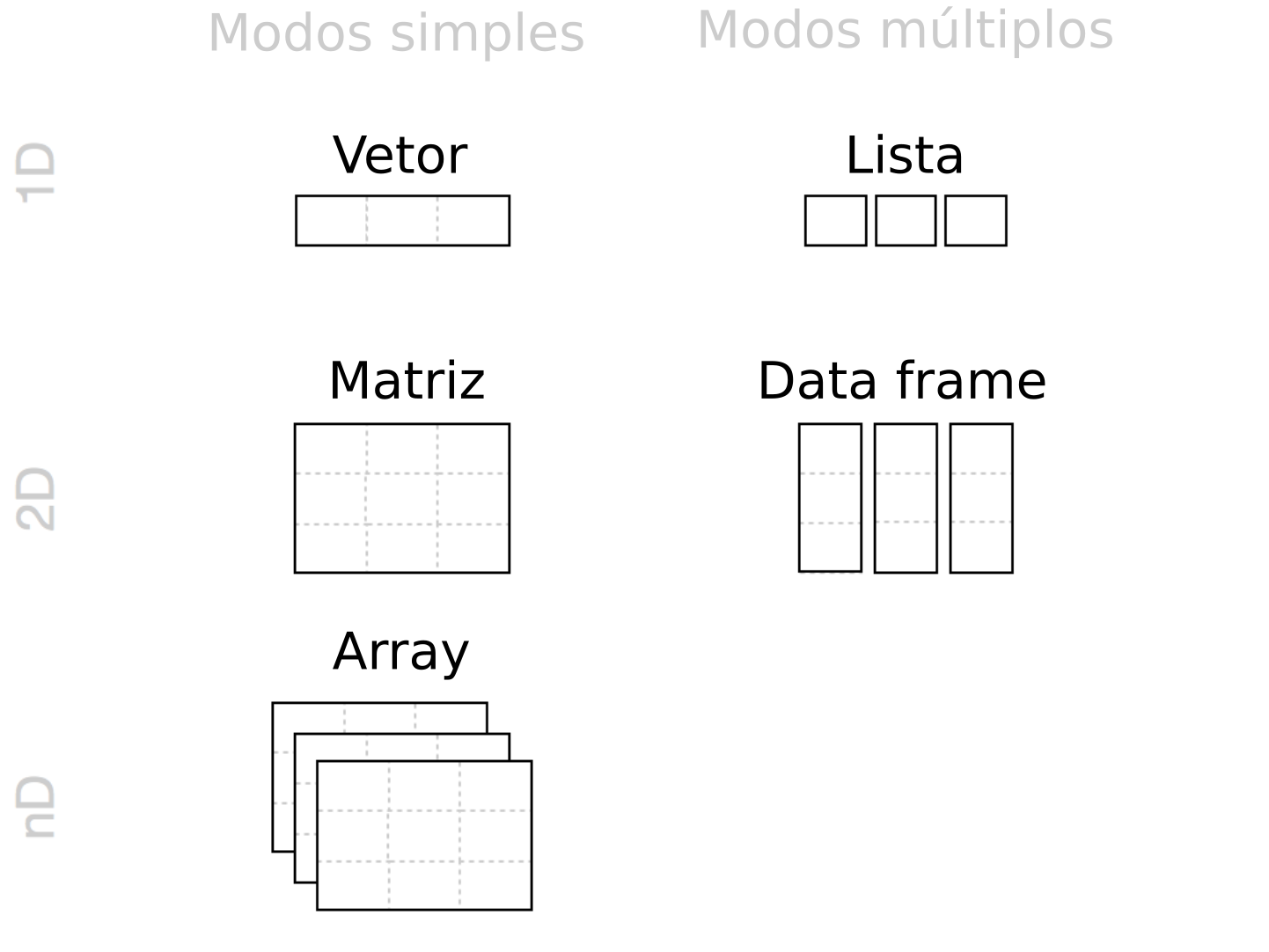
Figura 7.3: Estruturas de dados mais comuns no R: vetores, matrizes, arrays, listas e data frames. Adaptado de: R for Data Science (2e) .
Vetor
Vetores representam o encadeamento de elementos numa sequência unidimensional. Dessa forma, no R, essa estrutura de dados pode ser traduzida como medidas de uma variável numérica (discretas ou contínuas), variável binária (booleana - TRUE e FALSE) ou descrição (informações em texto).
Há diversas formas de se criar um vetor no R:
- Concatenando elementos com a função
c() - Criando sequências unitárias
:ou com a funçãoseq() - Criando repetições com a função
rep() - “Colar” palavras com uma sequência numérica com a função
paste()oupaste0() - Amostrando aleatoriamente elementos com a função
sample()
## [1] 15 18 20 22 18## [1] 1 2 3 4 5 6 7 8 9 10## Sequência com diferentes espaçamentos
sequencia_esp <- seq(from = 0, to = 100, by = 10)
sequencia_esp## [1] 0 10 20 30 40 50 60 70 80 90 100## [1] TRUE FALSE TRUE FALSE TRUE FALSE TRUE FALSE TRUE FALSE## [1] "amostra 1" "amostra 2" "amostra 3" "amostra 4" "amostra 5"## [1] 76 89 32 44 54 9 45 61 93 81Como os vetores são homogêneos, i.e., só comportam um modo, quando combinamos mais de um modo no mesmo objeto ocorre uma dominância de modos. Existe, dessa forma, uma coerção dos elementos combinados para que todos fiquem iguais. Essa dominância segue essa ordem:
DOMINANTEcharacter > double > integer > logicalRECESSIVO
Além disso, podemos utilizar as conversões listadas anteriormente para alterar os modos. Vamos exemplificar combinando os vetores criados anteriormente e convertendo-os.
## [1] "amostra 1" "amostra 2" "amostra 3" "amostra 4" "amostra 5" "76" "89"
## [8] "32" "44" "54" "9" "45" "61" "93"
## [15] "81"## [1] 1 0 1 0 1 0 1 0 1 0Fator
O fator representa medidas de uma variável categórica, podendo ser nominal ou ordinal. É fundamental destacar que fatores no R devem ser entendidos como um vetor de integer, i.e., ele é composto por números inteiros representando os níveis da variável categórica.
Para criar um fator no R usamos uma função específica factor(), na qual podemos especificar os níveis com o argumento level, ou fazemos uma conversão usando a função as.factor(). Trabalhar com fatores no R Base não é das tarefas mais agradáveis, sendo assim, no Capítulo 8 usamos a versão tidyverse usando o pacote forcats. Destacamos ainda a existência de fatores nominais para variáveis categóricas nominais e fatores ordinais para variáveis categóricas ordinais, quando há ordenamento entre os níveis, como dias da semana ou classes de altura.
## Fator nominal
fator_nominal <- factor(x = sample(x = c("floresta", "pastagem", "cerrado"),
size = 20, replace = TRUE),
levels = c("floresta", "pastagem", "cerrado"))
fator_nominal## [1] floresta pastagem pastagem pastagem floresta pastagem floresta pastagem floresta
## [10] cerrado floresta floresta cerrado floresta floresta cerrado cerrado floresta
## [19] floresta floresta
## Levels: floresta pastagem cerrado## Fator ordinal
fator_ordinal <- factor(x = sample(x = c("baixa", "media", "alta"),
size = 20, replace = TRUE),
levels = c("baixa", "media", "alta"), ordered = TRUE)
fator_ordinal## [1] baixa baixa alta media media alta alta media alta media baixa alta alta media
## [15] alta alta alta alta media media
## Levels: baixa < media < alta## Conversão
fator <- as.factor(x = sample(x = c("floresta", "pastagem", "cerrado"),
size = 20, replace = TRUE))
fator## [1] cerrado cerrado floresta floresta pastagem floresta cerrado cerrado floresta
## [10] pastagem pastagem pastagem cerrado pastagem floresta cerrado pastagem pastagem
## [19] pastagem pastagem
## Levels: cerrado floresta pastagemMatriz
A matriz representa dados no formato de tabela, com linhas e colunas. As linhas geralmente representam unidades amostrais (locais, transectos, parcelas) e as colunas representam variáveis numéricas (discretas ou contínuas), variáveis binárias (TRUE ou FALSE) ou descrições (informações em texto).
Podemos criar matrizes no R de duas formas. A primeira delas dispondo elementos de um vetor em um certo número de linhas e colunas com a função matrix(), podendo preencher essa matriz com os elementos do vetor por linhas ou por colunas alterando o argumento byrow.
## Vetor
ve <- 1:12
## Matrix - preenchimento por linhas - horizontal
ma_row <- matrix(data = ve, nrow = 4, ncol = 3, byrow = TRUE)
ma_row## [,1] [,2] [,3]
## [1,] 1 2 3
## [2,] 4 5 6
## [3,] 7 8 9
## [4,] 10 11 12## Matrix - preenchimento por colunas - vertical
ma_col <- matrix(data = ve, nrow = 4, ncol = 3, byrow = FALSE)
ma_col## [,1] [,2] [,3]
## [1,] 1 5 9
## [2,] 2 6 10
## [3,] 3 7 11
## [4,] 4 8 12A segundo forma, podemos combinar vetores, utilizando a função rbind() para combinar vetores por linha, i.e., um vetor embaixo do outro, e cbind() para combinar vetores por coluna, i.e., um vetor ao lado do outro.
## Criar dois vetores
vec_1 <- c(1, 2, 3)
vec_2 <- c(4, 5, 6)
## Combinar por linhas - vertical - um embaixo do outro
ma_rbind <- rbind(vec_1, vec_2)
ma_rbind## [,1] [,2] [,3]
## vec_1 1 2 3
## vec_2 4 5 6## vec_1 vec_2
## [1,] 1 4
## [2,] 2 5
## [3,] 3 6Array
O array representa combinação de tabelas, com linhas, colunas e dimensões. Essa combinação pode ser feita em múltiplas dimensões, mas apesar disso, geralmente é mais comum o uso em Ecologia para três dimensões, por exemplo: linhas (unidades amostrais), colunas (espécies) e dimensão (tempo). Isso gera um “cubo mágico” ou “cartas de um baralho”, onde podemos comparar, nesse caso, comunidades ao longo do tempo. Além disso, arrays também são muito comuns em morfometria geométrica ou sensoriamento remoto.
Podemos criar arrays no R dispondo elementos de um vetor em um certo número de linhas, colunas e dimensões com a função array(). Em nosso exemplo, vamos compor cinco comunidades de cinco espécies ao longo de três períodos.
## , , 1
##
## [,1] [,2] [,3] [,4] [,5]
## [1,] 1 0 0 0 1
## [2,] 0 1 0 1 0
## [3,] 0 1 1 1 1
## [4,] 1 0 1 0 0
## [5,] 1 1 1 1 1
##
## , , 2
##
## [,1] [,2] [,3] [,4] [,5]
## [1,] 0 1 1 0 1
## [2,] 0 1 0 1 1
## [3,] 1 0 0 0 1
## [4,] 0 1 0 0 0
## [5,] 0 1 1 1 0
##
## , , 3
##
## [,1] [,2] [,3] [,4] [,5]
## [1,] 1 1 1 1 0
## [2,] 0 1 1 1 1
## [3,] 0 1 1 1 0
## [4,] 0 1 0 0 0
## [5,] 0 0 1 1 0Data frame
O data frame também representa dados no formato de tabela, com linhas e colunas, muito semelhante à matriz. Mas diferentemente das matrizes, os data frames comportam mais de um modo em suas colunas. Dessa forma, as linhas do data frame ainda representam unidades amostrais (locais, transectos, parcelas), mas as colunas agora podem representar descrições (informações em texto), variáveis numéricas (discretas ou contínuas), variáveis binárias (TRUE ou FALSE) e variáveis categóricas (nominais ou ordinais).
A forma mais simples de se criar data frames no R é através da combinação de vetores. Essa combinação é feita com a função data.frame() e ocorre de forma horizontal, semelhante à função cbind(). Sendo assim, todos os vetores precisam ter o mesmo número de elementos, ou seja, o mesmo comprimento. Podemos ainda nomear as colunas de cada vetor. Outra forma, seria converter uma matriz em um data frame, utilizando a função as.data.frame().
## Criar três vetores
vec_ch <- c("sp1", "sp2", "sp3")
vec_nu <- c(4, 5, 6)
vec_fa <- factor(c("campo", "floresta", "floresta"))
## Data frame - combinar por colunas - horizontal - um ao lado do outro
df <- data.frame(vec_ch, vec_nu, vec_fa)
df## vec_ch vec_nu vec_fa
## 1 sp1 4 campo
## 2 sp2 5 floresta
## 3 sp3 6 floresta## Data frame - nomear as colunas
df <- data.frame(especies = vec_ch,
abundancia = vec_nu,
vegetacao = vec_fa)
df## especies abundancia vegetacao
## 1 sp1 4 campo
## 2 sp2 5 floresta
## 3 sp3 6 floresta## [,1] [,2] [,3]
## [1,] 1 2 3
## [2,] 4 5 6
## [3,] 7 8 9
## [4,] 10 11 12## V1 V2 V3
## 1 1 2 3
## 2 4 5 6
## 3 7 8 9
## 4 10 11 12Lista
A lista é um tipo especial de vetor que aceita objetos como elementos. Ela é a estrutura de dados utilizada para agrupar objetos, e é geralmente a saída de muitas funções.
Podemos criar listas através da função list(). Essa função funciona de forma semelhante à função c() para a criação de vetores, mas agora estamos concatenando objetos. Podemos ainda nomear os elementos (objetos) que estamos combinando.
Um ponto interessante para entender data frames, é que eles são listas, em que todos os elementos (colunas) possuem o mesmo número de elementos, ou seja, mesmo comprimento.
## Lista
lista <- list(rep(1, 20), # vector
factor(1, 1), # factor
cbind(c(1, 2), c(1, 2))) # matrix
lista## [[1]]
## [1] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
##
## [[2]]
## [1] 1
## Levels: 1
##
## [[3]]
## [,1] [,2]
## [1,] 1 1
## [2,] 2 2## Lista - nomear os elementos
lista_nome <- list(vector = rep(1, 20), # vector
factor = factor(1, 1), # factor
matrix = cbind(c(1, 2), c(1, 2))) # matrix
lista_nome## $vector
## [1] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
##
## $factor
## [1] 1
## Levels: 1
##
## $matrix
## [,1] [,2]
## [1,] 1 1
## [2,] 2 2Funções
Uma última estrutura de objetos criados no R são as funções. Elas são objetos criados pelo usuário e reutilizados para fazer operações específicas. A criação de funções geralmente é um tópico tratado num segundo momento, quando o usuário de R adquire certo conhecimento da linguagem. Aqui abordaremos apenas seu funcionamento básico, diferenciando sua estrutura para entendimento e sua diferenciação das demais estruturas.
Vamos criar uma função simples que retorna a multiplicação de dois termos. Criaremos a função com o nome multi, à qual será atribuída uma função com o nome function(), com dois argumentos x e y. Depois disso abrimos chaves {}, que é onde iremos incluir nosso bloco de código. Nosso bloco de código é composto por duas linhas, a primeira contendo a operação de multiplicação dos argumento com a atribuição ao objeto mu e a segunda contendo a função return() para retornar o valor da multiplicação.
## function(x, y){
##
## mu <- (x * y)
## return(mu)
##
## }## [1] 9667.4.2 Manipulação de objetos unidimensionais
Vamos agora explorar formas de manipular elementos de objetos unidimensionais, ou seja, vetores, fatores e listas.
A primeira forma de manipulação é através da indexação, utilizando os operadores []. Com a indexação podemos acessar elementos de vetores e fatores por sua posição. Utilizaremos números, sequência de números ou operações booleanas para retornar partes dos vetores ou fatores. Podemos ainda retirar elementos dessas estruturas com o operador aritmético -.
No exemplo a seguir, iremos fixar o ponto de partida da amostragem da função sample(), utilizando a função set.seed(42) (usamos 42 porque é a resposta para a vida, o universo e tudo mais - O Guia do Mochileiro das Galáxias, mas poderia ser outro número qualquer). Isso permite que o resultado da amostragem aleatória seja igual em diferentes computadores.
## Fixar a amostragem
set.seed(42)
## Amostrar 10 elementos de uma sequência
ve <- sample(x = seq(0, 2, .05), size = 10)
ve## [1] 1.80 0.00 1.20 0.45 1.75 0.85 1.15 0.30 1.90 0.20## [1] 1.75## [1] 1.80 0.00 1.20 0.45 1.75## [1] 1.80 0.00 1.20 0.45 1.75 0.85 1.15 0.30 1.90## [1] 1.8 0.2Podemos ainda fazer uma seleção condicional do vetor. Ao utilizarmos operadores relacionais, teremos como resposta um vetor lógico. Esse vetor lógico pode ser utilizado dentro da indexação para seleção de elementos.
## [1] TRUE FALSE TRUE FALSE TRUE FALSE TRUE FALSE TRUE FALSE## [1] 1.80 1.20 1.75 1.15 1.90Além da indexação, temos algumas funções que nos auxiliam em algumas operações com objetos unidimensionais.
| Função | Descrição |
|---|---|
max() |
Valor máximo |
min() |
Valor mínimo |
range() |
Amplitude |
length() |
Comprimento |
sum() |
Soma |
cumsum() |
Soma cumulativa |
prod() |
Produto |
sqrt() |
Raiz quadrada |
abs() |
Valor absoluto |
exp() |
Expoente |
log() |
Logaritmo natural |
log1p() |
Logaritmo natural mais 1 log(x + 1) |
log2() |
Logaritmo base 2 |
log10() |
Logaritmo base 10 |
mean() |
Média |
mean.weighted() |
Média ponderada |
var() |
Variância |
sd() |
Desvio Padrão |
mediam() |
Mediana |
quantile() |
Quantil |
quarters() |
Quartil |
IQR() |
Amplitude interquartil |
round() |
Arredondamento |
sort() |
Ordenação |
order() |
Posição ordenada |
rev() |
Reverso |
unique() |
Únicos |
summary() |
Resumo estatístico |
cut() |
Divide variável contínua em fator |
pretty() |
Divide variável contínua em intervalos |
scale() |
Padronização e centralização |
sub() |
Substitui caracteres |
grep() |
Posição de caracteres |
any() |
Algum valor? |
all() |
Todos os valores? |
which() |
Quais valores? |
subset() |
Subconjunto |
ifelse() |
Operação condicional |
Para listas, também podemos usar a indexação [] para acessar ou retirar elementos.
## $elem1
## [1] 1## $elem2
## [1] 2
##
## $elem3
## [1] 3Podemos ainda usar a indexação dupla [[]] para acessar os valores desses elementos.
## [1] 1## [1] 2Para listas nomeadas, podemos ainda utilizar o operador $ para acessar elementos pelo seu nome.
## [1] 1E ainda podemos utilizar funções para medir o comprimento dessa lista, listar os nomes dos elementos ou ainda renomear os elementos: length() e names().
## [1] 3## [1] "elem1" "elem2" "elem3"## $elemento01
## [1] 1
##
## $elemento02
## [1] 2
##
## $elemento03
## [1] 37.4.3 Manipulação de objetos multidimensionais
Da mesma forma que para objetos unidimensionais, podemos manipular elementos de objetos multidimensionais, ou seja, matrizes, data frames e arrays.
Novamente, a primeira forma de manipulação é através da indexação, utilizando os operadores []. Com a indexação podemos acessar elementos de matrizes, data frames e arrays por sua posição. Podemos ainda retirar elementos dessas estruturas com o operador aritmético -.
Entretanto, agora temos mais de uma dimensão na estruturação dos elementos dentro dos objetos. Assim, utilizamos números, sequência de números ou operação booleanas para retornar partes desses objetos, mas as dimensões têm de ser explicitadas e separadas por vírgulas para acessar linhas e colunas. Essa indexação funciona para matrizes e data frames. Para arrays, especificamos também as dimensões, também separadas por vírgulas para acessar essas dimensões.
## [,1] [,2] [,3]
## [1,] 1 5 9
## [2,] 2 6 10
## [3,] 3 7 11
## [4,] 4 8 12## [1] 3 7 11## [1] 5 6 7 8## [1] 5## [1] 1 5## [1] 1 9## [,1] [,2] [,3]
## [1,] 2 6 10
## [2,] 3 7 11
## [3,] 4 8 12## [,1] [,2]
## [1,] 1 5
## [2,] 2 6
## [3,] 3 7
## [4,] 4 8Para data frames, além de utilizar números e/ou sequências de números dentro do operador [] simples, podemos utilizar o operador [[]] duplo para retornar apenas os valores de uma linha ou uma coluna. Se as colunas estiverem nomeadas, podemos utilizar o nome da coluna de interesse entre aspas dentro dos operadores [] (retornar coluna) e [[]] (retornar apenas os valores), assim como ainda podemos utilizar o operador $ para data frames. Essas últimas operações retornam um vetor, para o qual podemos fazer operações de vetores ou ainda atualizar o valor dessa coluna selecionada ou adicionar outra coluna.
## Criar três vetores
sp <- paste("sp", 1:10, sep = "")
abu <- 1:10
flo <- factor(rep(c("campo", "floresta"), each = 5))
## data frame
df <- data.frame(sp, abu, flo)
df## sp abu flo
## 1 sp1 1 campo
## 2 sp2 2 campo
## 3 sp3 3 campo
## 4 sp4 4 campo
## 5 sp5 5 campo
## 6 sp6 6 floresta
## 7 sp7 7 floresta
## 8 sp8 8 floresta
## 9 sp9 9 floresta
## 10 sp10 10 floresta## [1] "sp1" "sp2" "sp3" "sp4" "sp5" "sp6" "sp7" "sp8" "sp9" "sp10"## flo
## 1 campo
## 2 campo
## 3 campo
## 4 campo
## 5 campo
## 6 floresta
## 7 floresta
## 8 floresta
## 9 floresta
## 10 floresta## [1] campo campo campo campo campo floresta floresta floresta floresta
## [10] floresta
## Levels: campo floresta## [1] "sp1" "sp2" "sp3" "sp4" "sp5" "sp6" "sp7" "sp8" "sp9" "sp10"## [1] 10## [1] "character"## Adicionar ou mudar colunas
set.seed(42)
df$abu2 <- sample(x = 0:1, size = nrow(df), rep = TRUE)
df## sp abu flo abu2
## 1 sp1 1 campo 0
## 2 sp2 2 campo 0
## 3 sp3 3 campo 0
## 4 sp4 4 campo 0
## 5 sp5 5 campo 1
## 6 sp6 6 floresta 1
## 7 sp7 7 floresta 1
## 8 sp8 8 floresta 1
## 9 sp9 9 floresta 0
## 10 sp10 10 floresta 1Podemos ainda fazer seleções condicionais para retornar linhas com valores que temos interesse, semelhante ao uso de filtro de uma planilha eletrônica.
## sp abu flo abu2
## 5 sp5 5 campo 1
## 6 sp6 6 floresta 1
## 7 sp7 7 floresta 1
## 8 sp8 8 floresta 1
## 9 sp9 9 floresta 0## sp abu flo abu2
## 6 sp6 6 floresta 1
## 7 sp7 7 floresta 1
## 8 sp8 8 floresta 1
## 9 sp9 9 floresta 0
## 10 sp10 10 floresta 1Além disso, há uma série de funções para conferência e manipulação de dados que listamos na Tabela seguinte:
| Função | Descrição |
|---|---|
head() |
Mostra as primeiras 6 linhas |
tail() |
Mostra as últimas 6 linhas |
nrow() |
Mostra o número de linhas |
ncol() |
Mostra o número de colunas |
dim() |
Mostra o número de linhas e de colunas |
rownames() |
Mostra os nomes das linhas (locais) |
colnames() |
Mostra os nomes das colunas (variáveis) |
str() |
Mostra as classes de cada coluna (estrutura) |
summary() |
Mostra um resumo dos valores de cada coluna |
rowSums() |
Calcula a soma das linhas (horizontal) |
colSums() |
Calcula a soma das colunas (vertical) |
rowMeans() |
Calcula a média das linhas (horizontal) |
colMeans() |
Calcula a média das colunas (vertical) |
table() |
Tabulação cruzada |
t() |
Matriz ou data frame transposto |
7.4.4 Valores faltantes e especiais
Valores faltantes e especiais são valores reservados que representam dados faltantes, indefinições matemáticas, infinitos e objetos nulos.
- NA (Not Available): significa dado faltante ou indisponível
- NaN (Not a Number): representa indefinições matemáticas
- Inf (Infinito): é um número muito grande ou um limite matemático
- NULL (Nulo): representa um objeto nulo, sendo útil para preenchimento em aplicações de programação
## var1 var2
## 1 1 1
## 2 4 4
## 3 2 5
## 4 NA 2## var1 var2
## [1,] FALSE FALSE
## [2,] FALSE FALSE
## [3,] FALSE FALSE
## [4,] TRUE FALSE## [1] TRUE## var1 var2
## 1 1 1
## 2 4 4
## 3 2 5## var1 var2
## 1 1 1
## 2 4 4
## 3 2 5
## 4 0 2## [1] 10## [1] NaN## Warning in log(-1): NaNs produced## [1] NaN## [1] Inf## [1] Inf## NULL7.4.5 Diretório de trabalho
O diretório de trabalho é o endereço da pasta (ou diretório) de onde o R importará ou exportar nossos dados.
Podemos utilizar o próprio RStudio para tal tarefa, indo em Session > Set Work Directory > Choose Directory... ou simplesmente utilizar o atalho Ctrl + Shift + H.
Podemos ainda utilizar funções do R para definir o diretório. Para tanto, podemos navegar com o aplicativo de gerenciador de arquivos (e.g., Windows Explorer) até nosso diretório de interesse e copiar o endereço na barra superior. Voltamos para o R e colamos esse endereço entre aspas como argumento da função setwd(). É fundamental destacar que no Windows é necessário inverter as barras (\ por / ou duplicar elas \\).
Aconselhamos ainda utilizar as funções getwd() para retornar o diretório definido na sessão do R, assim como as funções dir() ou list.files() para listagem dos arquivos no diretório, ambas medidas de conferência do diretório correto.
7.4.6 Importar dados
Uma das operações mais corriqueiras do R, antes de realizar alguma análise ou plotar um gráfico, é a de importar dados que foram tabulados numa planilha eletrônica e salvos no formato .csv, .txt ou .xlsx. Ao importar esse tipo de dado para o R, o formato que o mesmo assume, se nenhum parâmetro for especificado, é o da classe data frame, prevendo que a planilha de dados possua colunas com diferentes modos.
Existem diversas formas de importar dados para o R. Podemos importar utilizando o RStudio, indo na janela Environment e clicar em “Importar Dataset”.
Entretanto, aconselhamos o uso de funções que fiquem salvas em um script para aumentar a reprodutibilidade do mesmo. Dessa forma, as três principais funções para importar os arquivos nos três principais extensões (.csv, .txt ou .xlsx) são, respectivamente: read.csv(), read.table() e openxlsx::read.xlsx(), sendo o último do pacote openxlsx.
Para exemplificar como importar dados no R, vamos usar os dados de pinguins no Palmer Station, Antarctica Palmer Penguins. Faremos o download diretamente do site da fonte dos dados.
Vamos antes escolher um diretório de trabalho com a função setwd(), e em seguida criar um diretório com a função dir.create() chamado “dados”. Em seguida, vamos mudar nosso diretório para essa pasta e criar mais um diretório chamado “tabelas”, e por fim, definir esse diretório para que o conteúdo do download seja armazenado ali.
## Escolher um diretório
setwd("/home/mude/data/github/livro_aer")
## Criar um diretório 'dados'
dir.create("dados")
## Escolher diretório 'dados'
setwd("dados")
## Criar um diretório 'tabelas'
dir.create("tabelas")
## Escolher diretório 'tabelas'
setwd("tabelas")Agora podemos fazer o download do arquivo .csv e importar a tabela de dados.
## Download
download.file(url = "https://github.com/allisonhorst/palmerpenguins/blob/main/inst/extdata/penguins_raw.csv",
destfile = "penguins_raw.csv", method = "curl")Agora podemos importar a tabela de dados com a função read.csv(), atribuindo ao objeto penguins_raw.
Esse arquivo foi criado com separador de decimais sendo . e separador de colunas sendo ,. Caso tivesse sido criado com separador de decimais sendo , e separador de colunas sendo ;, usaríamos a função read.csv2().
Para outros formatos, basta usar as outras funções apresentadas, atentando-se para os argumentos específicos de cada função.
Caso o download não funcione ou haja problemas com a importação, os dados estao também no pacote palmerpenguins.
## Importar os dados pelo pacote palmerpenguins
pinguins_raw <- palmerpenguins::penguins_raw
head(pinguins_raw)## # A tibble: 6 × 17
## studyName `Sample Number` Species Region Island Stage `Individual ID` `Clutch Completion`
## <chr> <dbl> <chr> <chr> <chr> <chr> <chr> <chr>
## 1 PAL0708 1 Adelie … Anvers Torge… Adul… N1A1 Yes
## 2 PAL0708 2 Adelie … Anvers Torge… Adul… N1A2 Yes
## 3 PAL0708 3 Adelie … Anvers Torge… Adul… N2A1 Yes
## 4 PAL0708 4 Adelie … Anvers Torge… Adul… N2A2 Yes
## 5 PAL0708 5 Adelie … Anvers Torge… Adul… N3A1 Yes
## 6 PAL0708 6 Adelie … Anvers Torge… Adul… N3A2 Yes
## # ℹ 9 more variables: `Date Egg` <date>, `Culmen Length (mm)` <dbl>,
## # `Culmen Depth (mm)` <dbl>, `Flipper Length (mm)` <dbl>, `Body Mass (g)` <dbl>,
## # Sex <chr>, `Delta 15 N (o/oo)` <dbl>, `Delta 13 C (o/oo)` <dbl>, Comments <chr>7.4.7 Conferência dos dados importados
Uma vez importados os dados para o R, geralmente antes de iniciarmos qualquer manipulação, visualização ou análise de dados, fazemos a conferência desses dados. Dentre todas as funções de verificação, destacamos a importância destas funções apresentadas abaixo para saber se as variáveis foram importadas e interpretadas corretamente e reconhecer erros de digitação, por exemplo:
## # A tibble: 6 × 8
## species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g sex year
## <fct> <fct> <dbl> <dbl> <int> <int> <fct> <int>
## 1 Adelie Torgersen 39.1 18.7 181 3750 male 2007
## 2 Adelie Torgersen 39.5 17.4 186 3800 female 2007
## 3 Adelie Torgersen 40.3 18 195 3250 female 2007
## 4 Adelie Torgersen NA NA NA NA <NA> 2007
## 5 Adelie Torgersen 36.7 19.3 193 3450 female 2007
## 6 Adelie Torgersen 39.3 20.6 190 3650 male 2007## # A tibble: 6 × 8
## species island bill_length_mm bill_depth_mm flipper_length_mm body_mass_g sex year
## <fct> <fct> <dbl> <dbl> <int> <int> <fct> <int>
## 1 Chinstrap Dream 45.7 17 195 3650 female 2009
## 2 Chinstrap Dream 55.8 19.8 207 4000 male 2009
## 3 Chinstrap Dream 43.5 18.1 202 3400 female 2009
## 4 Chinstrap Dream 49.6 18.2 193 3775 male 2009
## 5 Chinstrap Dream 50.8 19 210 4100 male 2009
## 6 Chinstrap Dream 50.2 18.7 198 3775 female 2009## [1] 344## [1] 8## [1] 344 8## Nome das linhas e colunas
rownames(pinguins)
colnames(pinguins)
## Estrutura dos dados
str(pinguins)
## Resumo dos dados
summary(pinguins)
## Verificar NAs
any(is.na(pinguins))
which(is.na(pinguins))
## Remover as linhas com NAs
pinguins_na <- na.omit(pinguins) Importante
A função na.omit() retira a linha inteira que possui algum NA, inclusive as colunas que possuem dados que você não tem interesse em excluir. Dessa forma, tenha em mente quais dados você realmente quer remover da sua tabela.
Além das funções apresentadas, recomendamos olhar os seguintes pacotes que ajudam na conferência dos dados importados: Hmisc, skimr e inspectDF.
7.4.8 Exportar dados
Uma vez realizado as operações de manipulação ou tendo dados que foram analisados e armazenados num objeto no formato de data frame ou matriz, podemos exportar esses dados do R para o diretório que definimos anteriormente.
Para tanto, podemos utilizar funções de escrita de dados, como write.csv(), write.table() e openxlsx::write.xlsx(). Dois pontos são fundamentais: i) o nome do arquivo tem de estar entre aspas e no final dele deve constar a extensão que pretendemos que o arquivo tenha, e ii) é interessante utilizar os argumentos row.names = FALSE e quote=FALSE, para que o arquivo escrito não tenha o nome das linhas ou aspas em todas as células, respectivamente.
## Exportar dados na extensão .csv
write.csv(pinguins_na, "pinguins_na.csv",
row.names = FALSE, quote = FALSE)
## Exportar dados na extensão .txt
write.table(pinguins_na, "pinguins_na.txt",
row.names = FALSE, quote = FALSE)
## Exportar dados na extensão .xlsx
openxlsx::write.xlsx(pinguins_na, "pinguins_na.xlsx",
row.names = FALSE, quote = FALSE)7.5 Para se aprofundar
Listamos a seguir livros e links com material que recomendamos para seguir com sua aprendizagem em R Base.
7.5.1 Livros
Recomendamos aos (às) interessados(as) os livros: i) Crawley The R Book, ii) Davies The Book of R: A First Course in Programming and Statistics, iii) Gillespie e Lovelace Efficient R programming, iv) Holmes e Huber Modern Statistics for Modern Biology, v) Irizarry e Love Data Analysis for the Life Sciences with R, vi) James e colaboradores An Introduction to Statistical Learning: with Applications in R, vii) Kabacoff R in Action: Data analysis and graphics with R, viii) Matloff The Art of R Programming: A Tour of Statistical Software Design, ix) Long e Teetor R Cookbook e x) Wickham Advanced R.
7.5.2 Links
Existem centenas de ferramentas online para aprender e explorar o R. Dentre elas, indicamos os seguintes links (em português e inglês):
Introdução ao R
- An Introduction to R - Douglas A, Roos D, Mancini F, Couto A, Lusseau D
- A (very) shortintroduction to R - Paul Torfs & Claudia Brauer
- R for Beginners - Emmanuel Paradis
Ciência de dados - Ciência de Dados em R - Curso-R - Data Science for Ecologists and Environmental Scientists - Coding Club
Estatística - Estatística Computacional com R - Mayer F. P., Bonat W. H., Zeviani W. M., Krainski E. T., Ribeiro Jr. P. J - Data Analysis and Visualization in R for Ecologists - Data Carpentry
Miscelânea
7.6 Exercícios
4.1
Use o R para verificar o resultado da operação 7 + 7 ÷ 7 + 7 x 7 - 7.
4.2
Verifique através do R se 3x2³ é maior que 2x3².
4.3
Crie dois objetos (qualquer nome) com os valores 100 e 300. Multiplique esses objetos (função prod()) e atribuam ao objeto mult. Faça o logaritmo natural (função log()) do objeto mult e atribuam ao objeto ln.
4.4
Quantos pacotes existem no CRAN nesse momento? Execute essa combinação no Console: nrow(available.packages(repos = "http://cran.r-project.org")).
4.5
Instale o pacote tidyverse do CRAN.
4.6
Escolha números para jogar na mega-sena usando o R, nomeando o objeto como mega. Lembrando: são 6 valores de 1 a 60 e atribuam a um objeto.
4.7
Crie um fator chamado tr, com dois níveis (“cont” e “trat”) para descrever 30 locais de amostragem, 15 de cada tratamento. O fator deve ser dessa forma cont, cont, cont, ...., cont, trat, trat, ...., trat.
4.8
Crie uma matriz chamada ma, resultante da disposição de um vetor composto por 300 valores aleatórios entre 0 e 10. A matriz deve conter 30 linhas e ser disposta por colunas.
4.9
Crie um data frame chamado df, resultante da composição desses vetores:
id: 1:30sp: sp01, sp02, ..., sp29, sp30ab: 30 valores aleatórios entre 0 a 5
4.10
Crie uma lista com os objetos criados anteriormente: mega, tr, ma e df.
4.11
Selecione os elementos ímpares do objeto tr e atribua ao objeto tr_impar.
4.12
Selecione as linhas com ids pares do objeto df e atribua ao objeto df_ids_par.
4.13
Faça uma amostragem de 10 linhas do objeto df e atribua ao objeto df_amos10. Use a função set.seed() para fixar a amostragem.
4.14
Amostre 10 linhas do objeto ma, mas utilizando as linhas amostradas do df_amos10 e atribua ao objeto ma_amos10.
4.15
Una as colunas dos objetos df_amos10 e ma_amos10 e atribua ao objeto dados_amos10.